PROGRAME UTILIZATE IN CALCULUL INDICILOR TOPOLOGICI: ASEMANARI SI DEOSEBIRI
Numarul mare de descriptori moleculari din literatura de specialitate precum si structurile chimice cat mai complicate au impus scrierea si utilizarea unor programe software care sa calculeze acesti descriptori moleculari si in felul acesta sa reduca timpul si munca cercetatorilor. Printre cele mai utilizate pachete de programe sunt amintite: CODESSA (COmprehensive DEscriptors for Structural and Statistical Analysis), PRECLAV (PRoperty Evaluation by CLAss Variables) sau DRAGON. In cele ce urmeaza vor fi prezentate pe scurt cele trei programe precum si asemanarile sau deosebirile dintre acestea.
CODESSA este un program de predictie si analiza statistica fiind rezultatul efortului comun al companiei Semichem [286] si Profesorilor Alan Katritzky (Universitatea din Florida) si Mati Karelson (Universitatea Tartu, Estonia) [287-291].
Acest program calculeaza peste 450 de 15415y241p descriptori moleculari pe baza structurii geometrice sau electronice a moleculei. Acesti descriptori moleculari pot fi clasificati in sase grupe: constitutionali, topologici, geometrici, electrostatici, cuanto-chimici si
termodinamici. Diferite atribute structurale ale moleculei pot fi utilizate ca descriptori moleculari si anume: indici topologici de conectivitate (proprietati depinzand de distributia de sarcina in molecula), functii termodinamice la diferite temperaturi sau caracteristici de solvatare. Pe langa descriptorii deja definiti in literatura de specialitate in program mai pot fi introdusi si alti descriptori cu ajutorul operatiilor si functiilor matematice uzuale.
CODESSA utilizeaza mai multe tehnici de analiza statistica in corelarea datelor experimentale cu descriptorii moleculari calculati. O prima categorie o reprezinta analiza mono sau bidimensionala de distributie normala a datelor initiale sau studiul de intercorelare a diferitelor proprietati sau descriptori. O a doua categorie de analize statistice este reprezentata de metodele de regresie liniara sau multiliniara. Sunt de asemenea utilizate mai multe strategii heuristice pentru gasirea celor mai bune corelatii multiparametru in spatiul natural sau extins al descriptorilor.
Proprietatile moleculare, descriptorii sau combinatii ale acestora pot fi analizate utilizand factorul de analiza bazat pe metode de recunoastere structurala incluzand analiza componentului principal (PCA), cele mai mici patrate partiale neliniare (NIPALS) si transformarea tinta (TT).
CODESSA suporta urmatoarele tipuri de formate ca fisiere input: formatul MDL-molfile [292], formatul Hyperchem-hin [77], formatul Sybyl-mol [293] si doua formate AMPAC (rezultatul calculelor SCF si a calculelor termodinamice)-out. Primele trei formate codifica informatii privind constitutia atomica, conectivitatea si geometria 3D a structurilor chimice in timp ce formatele AMPAC codifica proprietatile cuanto-chimcie sau termodinamice ale structurilor chimice [286]. Fisierele output cuprind date numerice, grafice despre structuri, descriptori si proprietati precum si rezultatul analizei statistice.
AMPAC este primul pachet de programe semiempirice suportat de CODESSA care codifica informatii necesare calculului descriptorilor cuanto-chimici si termodinamici. Aceleasi rezultate se obtin si prin utilizarea ultimei versiuni a unui alt pachet de programe semiempirice - MOPAC [74].
In continuare sunt prezentate etapele de obtinere a fisierului output in cazul etanului care urmeaza a fi utilizat de CODESSA pentru calculul descriptorilor moleculari implemetati in acest program.
1. Modelarea si optimizarea geometriei etanului cu ajutorul programului Hiperchem [77] utilizandu-se campul de forte mecanice moleculare MM+ (Figura 76).
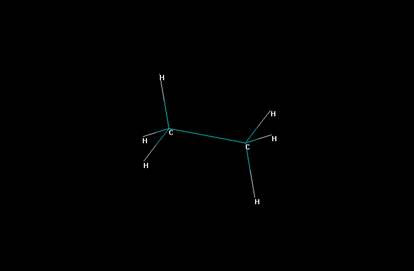
Figura 76. Graful molecular caracteristic etanului modelat si optimizat cu ajutorul programului Hiperchem [77] utilizandu-se campul de forte mecanice moleculare MM+
Fisierul etan.hin are urmatoarea compozitie
forcefield mm+
sys 0 0 1
view 40 0.38071 55 15 -0.2023222 -0.9449037 0.2573377 -0.9792282 0.1916154 -0.06629988 0.01333715 -0.2654062 -0.9640444 6.435e-005 -0.44528 -54.877
seed -1109
mol 1
atom 1 - C C4 - 0 -0.4522884 -0.6723938 1.663498e-006 4 2 s 3 s 4 s 5 s
atom 2 - C C4 - 0 -0.4522918 0.8594166 1.660006e-006 4 1 s 6 s 7 s 8 s
atom 3 - H H - 0 0.5883118 -1.072138 -2.855648e-006 1 1 s
atom 4 - H H - 0 -0.9725845 -1.07214 0.9011894 1 1 s
atom 5 - H H - 0 -0.9725913 -1.072142 -0.9011812 1 1 s
atom 6 - H H - 0 -1.492892 1.259161 -2.834491e-006 1 2 s
atom 7 - H H - 0 0.06800444 1.259163 0.9011894 1 2 s
atom 8 - H H - 0 0.06801117 1.259165 -0.9011812 1 2 s
endmol 1
2. Conversia extensiei hin din fisierul etan.hin in mpn cu ajutorul programului BabelWin [78] (etan.mpn).
In aceasta situatie fisierul etan.mpn are urmatoarea compozitie:
THERMO ROT=1 GEO-OK mmok precise CHARGE=0 GNORM=0.1 BONDS VECTORS DENSITY
C:\Proba\etan.mpn
C:\Proba\etan.mpn
C 0.0000000 0 0.000000 0 0.000000 0 0 0 0 0.0000
C 1.5318104 1 0.000000 0 0.000000 0 1 0 0 0.0000
H 1.1147395 1 111.014293 1 0.000000 0 1 2 0 0.0000
H 1.1147396 1 111.014210 1 120.000058 1 1 2 3 0.0000
H 1.1147396 1 111.014321 1 -120.000019 1 1 2 3 0.0000
H 1.1147396 1 111.014302 1 179.974377 1 2 1 3 0.0000
H 1.1147397 1 111.014218 1 -60.000433 1 2 1 3 0.0000
H 1.1147397 1 111.014330 1 59.999480 1 2 1 3 0.0000
Pentru etapele urmatoare fisierul etan.mpn va contine urmatoarele cuvinte cheie: THERMO ROT=1 GEO-OK mmok precise CHARGE=0 GNORM=0.1 BONDS VECTORS DENSITY a caror semnificatie este detaliata in referinta [74].
3. Obtinerea unei matrici .dat utilizand programul WinMopac [75].
Fisierul etan.dat are urmatoarea compozitie
CODESSA AM1 PRECISE GRAD GEO-OK LICENSE SYBYL SIZES TRUSTE RHF
C 0.0000000 0 0.000000 0 0.000000 0 0 0 0 0.0166
C 1.5209583 1 0.000000 0 0.000000 0 1 0 0 0.0166
H 1.1091640 1 111.190673 1 0.000000 0 1 2 0 -0.0055
H 1.1091427 1 111.197731 1 120.013740 1 1 2 3 -0.0055
H 1.1091841 1 111.181407 1 -119.986539 1 1 2 3 -0.0056
H 1.1091680 1 111.188812 1 179.998897 1 2 1 3 -0.0055
H 1.1092042 1 111.170851 1 -60.025821 1 2 1 3 -0.0056
H 1.1091349 1 111.200783 1 59.976585 1 2 1 3 -0.0055
Pentru etapele urmatoare fisierul etan.dat va contine urmatoarele cuvinte cheie: CODESSA AM1 PRECISE GRAD GEO-OK LICENSE SYBYL SIZES TRUSTE RHF a caror semnificatie este detaliata tot in referinta [75].
4. Obtinerea fisierului cu calcule cuanto-chimice si termochimice utilizand pachetul de programe semiempirice AMPAC [286].
Compozitia fisierului etan.out este este prezentata in Anexa 1 a prezentei teze de doctorat.
Fisierul etan.out este utilizat apoi de CODESSA pentru calculul descriptorilor moleculari generandu-se fisierul etan.cod care va contine date numerice, grafice despre etan, descriptori si proprietati precum si rezultatul analizei statistice.
In Figura 77 este prezentata sinteaza etapelor 1-4.
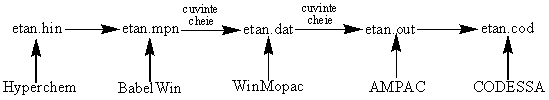
Figura 77. Sinteaza etapelor de obtinere a fisierului utilizat de CODESSA pentru calculul descriptorilor moleculari
PRECLAV PRoperty Evaluation by CLAss Variables) este un alt pachet software utilizat in calcule QSAR/QSPR fiind dezvoltat de Laszlo Tarko de la Centrul de Chimie Organica ,,C. D. Nenitescu'' al Academiei Romane, Bucuresti [294-295].
Pentru obtinerea fisierului utilizat de PRECLAV, moleculele sunt modelate utilizand programul de mecanica moleculara PCMODEL [296] urmand ca geometria si conformerii de minima energie sa se obtina utilizand campul de forte MMX si algoritmul GMMX [297]. Geometria este mai riguros optimizata cu ajutorul programului de mecanica cuantica MOPAC [74] utilizand cuvintele cheie: AM1 PULAY GNORM=0.01 SHIFT=50 GEO-OK MMOK CAMP-KING BONDS VECTORS.
Fisierul output creat de MOPAC este utilizat ca fisier input de PRECLAV care calculeaza peste 1100 de descriptori (globali, locali sau grid / field) urmand a se realiza si analiza statistica. PRECLAV este capabil sa calculeze si aromaticitatea legaturilor chimice pe baza algoritmlui TPA sau valoarea optima a distantei unor atomi proba.
Studiile QSAR se pot realiza cu sau fara set de testare. In cazul in care studiile QSAR se realizeaza cu un set de testare, acest program utilizeaza functia CLASS pentru identificarea celor mai semnificativi descriptori.
Ecuatiile QSAR ,,bune'' sunt selectate de program conform functiei de cross-validare.
DRAGON este un pachet software dezvoltat de Milano chemometrics [298] si Grupul de cercetare QSAR condus de Roberto Todeschini [299]. Acest program nu realizeaza optimizarea geometriei si nu transforma structurile topologice in structuri geometrice 3D corespunzatoare.
DRAGON calculeaza peste 1664 de descriptori clasificati in 20 de categorii. Pe langa descriptorii moleculari clasici sunt calculate si o serie de proprietati moleculare cum ar fi: logP, refractia molara, numarul de legaturi rotabile, legaturi H-acceptoare, H-donoare, regula Lipinski sau o serie de indici de toxicitate. Acest program accepta urmatoarele date de input: 1. Sybyl-TriposΘMol2 file(.mol, .ml2, .mol2); 2. Sybyl-CambridgeSoft Corp.ΘMolfiles (.sm2); 3. Sybyl-TriposΘ Multiple Molfiles(.mol, .ml2); 4. Molfiles-Molecular Design Ltd. (MDL)ΘMolfiles (.mol); 5. Multiple SD files- Molecular Design Ltd. (MDL)ΘMultiple SD files (.sdf); 6. Hyperchem-HypercubeΘFiles (.hin); 7. Smiles notations (.smi); 8. Macromodel-SchrodingerΘFiles(.dat, .out).
Fisierul output creat de Dragon contine valorile tuturor descriptorilor si este utilizat pentru realizarea unei statistici univariate sau a unor histograme.
Principalele diferente intre CODESSA si PRECLAV sunt urmatoarele:
a) 50% din descriptorii moleculari calculate de PRECLAV difera de cei calculate de CODESSA.
b) Programul PRECLAV calculeaza si utilizeaza descriptori grid-field in timp ce CODESSA nu utilizeaza asemenea descriptori.
c) Pentru selectia descriptorilor programul PRECLAV utilizeaza functia CLASS in loc de criteriile utilizate de CODESSA (numai cand se lucreaza cu seturi de testare).
d) In cazul programului PRECLAV selectia ecuatiilor QSAR se face in functie de valoarea functiei de cross-validare in timp ce CODESSA nu utilizeaza acest criteriu.
e) Programul PRECLAV utilizeaza ca metoda statistica numai BMLR (Best MultiLinear Regression) in timp ce CODESSA utilizeaza si alte metode statistice (metoda heuristica, PLS, PCA).
Diferenta majora dintre CODESSA, PRECLAV si DRAGON este ca DRAGON calculeaza un numar mult mai mare de descriptori precum si serie de proprietati moleculare cum ar fi: logP, refractia molara, numarul de legaturi rotabile, legaturi H-acceptoare, H-donoare, regula Lipinski sau indici de toxicitate dar analiza statistica nu este la nivelul primelor doua programe.
|